Bùi Minh Nhật
Intern Writer
Các nhà khoa học tại Phòng thí nghiệm quốc gia Oak Ridge (ORNL) thuộc Bộ Năng lượng Mỹ vừa phát triển một công cụ mạnh mẽ, kết hợp trí tuệ nhân tạo (AI) với mô phỏng động lực học phân tử, giúp dự đoán chính xác cách thực vật và vi khuẩn giao tiếp, hình thành mối quan hệ cộng sinh ngay từ cấp độ cơ bản nhất.
Quy trình làm việc mới này cho phép các nhà nghiên cứu xác định những gen của thực vật đóng vai trò quan trọng trong việc kiểm soát mối quan hệ cộng sinh với vi sinh vật. Nhờ đó, họ có thể thiết kế những “hệ vi sinh vật hỗ trợ” giúp cây trồng phát triển nhanh hơn, giảm lượng phân bón cần dùng và tạo ra nhiều sinh khối hơn để chuyển hóa thành nhiên liệu, hóa chất hoặc vật liệu giá trị. Đây được xem là một hướng đi tiềm năng, thúc đẩy an ninh lương thực năng lượng và tăng sức cạnh tranh của Mỹ trong lĩnh vực công nghệ sinh học toàn cầu.
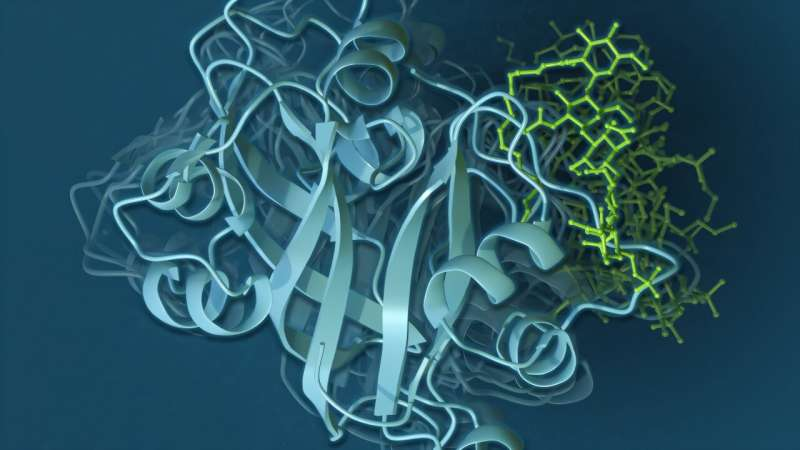
Trong tự nhiên, thực vật và vi sinh vật giao tiếp thông qua các tín hiệu hóa học gọi là phối tử. Một nhóm phối tử đặc biệt, lipo-chitooligosaccharides (LCO), đóng vai trò then chốt trong việc giúp rễ cây và vi khuẩn cộng sinh, cải thiện sức khỏe và sự phát triển của cây trồng. Tuy nhiên, việc dự đoán protein nào trong tế bào có thể nhận biết và liên kết với các LCO vẫn là bài toán khó do cấu trúc lớn và linh hoạt của chúng.
Các công cụ tính toán phổ biến như AlphaFold vốn dự đoán hình dạng ba chiều của protein chỉ hỗ trợ một phần, bởi chúng chủ yếu được huấn luyện trên dữ liệu về các phối tử nhỏ hơn, và thường bỏ qua các chuyển động liên tục của những phân tử phức tạp như LCO.
Cách tiếp cận MD/ML này giúp xếp hạng mức độ liên kết giữa các thụ thể thực vật và phối tử, kể cả khi cấu trúc protein ban đầu chỉ là những mô hình thô. Kết quả thu được phù hợp với các thí nghiệm trong phòng lab, đồng thời tiết lộ nhiều chi tiết mới về cách những phân tử này tương tác.
Theo Erica Prates, đồng trưởng nhóm dự án, việc nhanh chóng dự đoán các “cặp đôi” phân tử giúp các nhà khoa học tập trung vào những thí nghiệm quan trọng, tiết kiệm thời gian và chi phí. Omar Demerdash, một đồng tác giả khác, cho biết cường độ liên kết giữa phối tử và thụ thể quyết định các gen nào được kích hoạt cũng như nhiều quá trình sinh lý khác.
Dan Jacobson, nhà sinh học hệ thống tại ORNL, nhấn mạnh: “Protein không hề cứng nhắc, chúng luôn dao động. Khi đưa yếu tố chuyển động vào mô phỏng, chúng ta có được bức tranh toàn diện hơn về cách chúng liên kết với phối tử”. Ông cho rằng phương pháp này không chỉ giúp hiểu sâu hơn về sự cộng sinh thực vật vi sinh vật mà còn có thể hỗ trợ phát hiện hướng tái sử dụng thuốc trong điều trị các bệnh lý ở người.
Với sự kết hợp của AI và mô phỏng phân tử, nghiên cứu tại ORNL mở ra triển vọng đẩy nhanh quá trình chọn lọc vi sinh vật có lợi, hỗ trợ cây trồng khỏe mạnh hơn và góp phần thúc đẩy các giải pháp bền vững trong nông nghiệp lẫn y học. (phys)
Quy trình làm việc mới này cho phép các nhà nghiên cứu xác định những gen của thực vật đóng vai trò quan trọng trong việc kiểm soát mối quan hệ cộng sinh với vi sinh vật. Nhờ đó, họ có thể thiết kế những “hệ vi sinh vật hỗ trợ” giúp cây trồng phát triển nhanh hơn, giảm lượng phân bón cần dùng và tạo ra nhiều sinh khối hơn để chuyển hóa thành nhiên liệu, hóa chất hoặc vật liệu giá trị. Đây được xem là một hướng đi tiềm năng, thúc đẩy an ninh lương thực năng lượng và tăng sức cạnh tranh của Mỹ trong lĩnh vực công nghệ sinh học toàn cầu.

Trong tự nhiên, thực vật và vi sinh vật giao tiếp thông qua các tín hiệu hóa học gọi là phối tử. Một nhóm phối tử đặc biệt, lipo-chitooligosaccharides (LCO), đóng vai trò then chốt trong việc giúp rễ cây và vi khuẩn cộng sinh, cải thiện sức khỏe và sự phát triển của cây trồng. Tuy nhiên, việc dự đoán protein nào trong tế bào có thể nhận biết và liên kết với các LCO vẫn là bài toán khó do cấu trúc lớn và linh hoạt của chúng.
Các công cụ tính toán phổ biến như AlphaFold vốn dự đoán hình dạng ba chiều của protein chỉ hỗ trợ một phần, bởi chúng chủ yếu được huấn luyện trên dữ liệu về các phối tử nhỏ hơn, và thường bỏ qua các chuyển động liên tục của những phân tử phức tạp như LCO.
Phương pháp MD/ML: Rút ngắn thời gian nghiên cứu và mở ra ứng dụng mới
Để khắc phục hạn chế trên, nhóm nghiên cứu tại ORNL đã phát triển phương pháp lai mới, kết hợp mô phỏng động lực học phân tử (MD) với học máy (ML). Họ huấn luyện mô hình trên một kho dữ liệu lớn về các phức hợp protein phối tử, rồi chạy các mô phỏng trên hai siêu máy tính Frontier và Summit tại Cơ sở máy tính lãnh đạo Oak Ridge.Cách tiếp cận MD/ML này giúp xếp hạng mức độ liên kết giữa các thụ thể thực vật và phối tử, kể cả khi cấu trúc protein ban đầu chỉ là những mô hình thô. Kết quả thu được phù hợp với các thí nghiệm trong phòng lab, đồng thời tiết lộ nhiều chi tiết mới về cách những phân tử này tương tác.
Theo Erica Prates, đồng trưởng nhóm dự án, việc nhanh chóng dự đoán các “cặp đôi” phân tử giúp các nhà khoa học tập trung vào những thí nghiệm quan trọng, tiết kiệm thời gian và chi phí. Omar Demerdash, một đồng tác giả khác, cho biết cường độ liên kết giữa phối tử và thụ thể quyết định các gen nào được kích hoạt cũng như nhiều quá trình sinh lý khác.
Dan Jacobson, nhà sinh học hệ thống tại ORNL, nhấn mạnh: “Protein không hề cứng nhắc, chúng luôn dao động. Khi đưa yếu tố chuyển động vào mô phỏng, chúng ta có được bức tranh toàn diện hơn về cách chúng liên kết với phối tử”. Ông cho rằng phương pháp này không chỉ giúp hiểu sâu hơn về sự cộng sinh thực vật vi sinh vật mà còn có thể hỗ trợ phát hiện hướng tái sử dụng thuốc trong điều trị các bệnh lý ở người.
Với sự kết hợp của AI và mô phỏng phân tử, nghiên cứu tại ORNL mở ra triển vọng đẩy nhanh quá trình chọn lọc vi sinh vật có lợi, hỗ trợ cây trồng khỏe mạnh hơn và góp phần thúc đẩy các giải pháp bền vững trong nông nghiệp lẫn y học. (phys)